Vector y dataframe.
Paquetes Necesarios#
install.packages("rmarkdown")
install.packages("dplyr")
install.packages("reshape2")
install.packages("remotes")
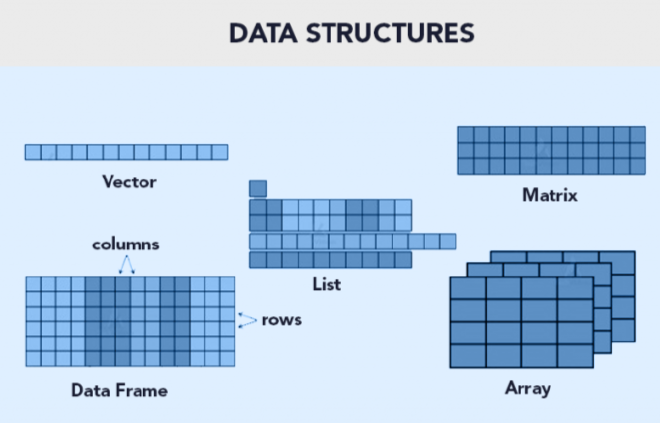
Estructura de Datos/Tipos de Datos#
Existen 5 estructuras de datos en R#
Las estructuras de datos son objetos que contienen datos. Cuando trabajamos con R, lo que estamos haciendo es manipular estas estructuras.
| TIpo | Declaracion de la variable | Conversion | Dimensiones | Contenido |
|---|---|---|---|---|
| Vector | c(), seq(), rep(), valorI:valorN; seq_len() | as.vector() | 1 | homogenea |
| Lists | list() | as.list() | 1* | homogenea |
| Matrix | matrix(valores, nrow = x, ncol = y) | as.matrix() | 2 | heterogenea |
| Dataframe | data.frame() | as.dataframe() | 2 | heterogenea |
Tambien estan los array pero son poco usados en R.
Para conocer la estructura del archivo debes usar str().
Una lista puede contener matrices en su interior, vectores, funciones, etc.
| Clases | Declaración de la variable | Conversión de formato | Reglas |
|---|---|---|---|
| numeric | numeric() | as.numeric() | FALSE -> 0, TRUE -> 1; “1”, “2”, … -> 1,2, …; “A” -> NA |
| integer | integer() | as.integer() | FALSE -> 0, TRUE -> 1; “1”, “2”, … -> 1,2, …; “A” -> NA |
| double | double() | as.double() | |
| character | character() | as.character() | 1,2, … -> “1”, “2”, …; FALSE -> “FALSE”; TRUE -> “TRUE” |
| logical | logical() | as.logical() | 0 -> FALSE, other numbers -> TRUE; (“FALSE”, “F”) -> FALSE; (“TRUE”, “T”) -> TRUE, other character -> NA |
| factor | factor() | as.factor() |
Si queremos saber la clase de la variable debemos usar class(), aunque tambien str() te dice esta información.
NOTA:
as.integer()convierte los numeros a enteros. Ejemplo: as.integer(2.5) = 2. En cambio,as.double()permite que los numeros contengan decimales.
Recordatorio: Operadores#
| Aritméticos | Comparación | Argumentos lógicos (Logical Operators) |
|---|---|---|
+ Adición / suma | < Menor que | !x - Not x (logical NOT) |
- Sustracción / resta | > Mayor que | x & y OR x AND y - (logical AND) |
* Multiplicación | <= Menor o igual que | x && y - identico |
/ OR %% División | >= Mayor o igual que | xor(x,y) Funcion OR |
^ OR ** Exponencial | == Igual a | %in% pertenece a |
%/% División integral | != Diferente de |
Otra forma de escrir OR es x | y OR x || y.
Jerarquía de operaciones#
En R, al igual que en matemáticas, las operaciones tienen un orden de evaluación definido.
Cuanto tenemos varias operaciones ocurriendo al mismo tiempo, en realidad, algunas de ellas son realizadas antes que otras y el resultado de ellas dependerá de este orden.
| Orden | Operadores |
|---|---|
| 1 | ^ |
| 2 | * / |
| 3 | + - |
| 4 | < > <= >= == != |
| 5 | ! |
| 6 | & |
| 7 | OR |
Manipulación de datos con R base (vector y dataframe)#
Vector: Formas de declarar un vector#
# Declarar el vector con 5 posiciones (del 1 al 5)
# Opcion A - c()
x <- c(1,2,3,4,5)
# Opcion B - valorI:valorN
x <- 1:5
x
# Opcion C - seq(inicio, final)
x <- seq(1,5)
x
# Opcion D -seq_len(final)
x <- seq_len(5)
x
\# Opcion E - rep(valor, veces)
y <- c(rep(1,5), rep(c(3,5),5))
y
Vector: Estructura y clases de variables#
Conocer la estructura de un vector mediante la función str(). El vector es de tipo integer y va de 1 a 5.
str(x)
Para solo ver el tipo usamos la función class()
class(x)
Ejercicios#
x <- c(TRUE, TRUE, FALSE, FALSE)
x
x <- as.factor(x)
Vamos a crear un nuevo vector con los nombres de los participantes a esta clase
ClaseBioinfo <- c("Jose Luis", "Diego", "Rosita", "Yuri", "Ulises", "Tere", "Chucho")
ClaseBioinfo
#ver estructura
str(ClaseBioinfo)
Extraer información
ClaseBioinfo =="Ulises"
ClaseBioinfo =="Chucho"
Cuando son mas de un valor usamos %in%
ClaseBioinfo %in% c("Ulises", "Chucho")
Ahora tambien podemos usar:
ClaseBioinfo == c("Ulises", "Chucho")
Podemos declarar un vector de los tratamientos
#Opcion A
y <- rep(c("Control", "Condicion"),5)
#Opcion B
y <- c(rep("Control",5), rep("Condicion",5))
y
Dataframe#
Dataframe: Formas de declarar un vector#
x <- 1:4
n <- 10
M <- c(10, 35)
y <- 2:4
Podemos declarar los componentes de un Dataframe en variables separadas y luego juntarlo o en una sola linea.
x <- 1:4; n <- 10; M <- c(10, 35); y <- 2:4
Vamos a crear dataframes apartir de estos vectores.
data.frame(x,n)
data.frame(x, M)
Dataframe en una sola linea#
data.frame(x = 1:4, n = 10)
data.frame(x = 1:4, M = c(10,35))
Dataframe: Ejercicio#
data.frame(x, y)
NOTA: El número de filas y columnas debe ser el mismo número. Existen excepciones al usar paquetes como dplyr.
Dataframe: Estructura y clases de variables#
Cada columna es una variable la cual puede ser de un tipo o clase.
df <- data.frame(genes = paste0("Gen", seq_len(8)),
expression = c(3.8, 5.5, 6.3, 1.8, 9, rep(3,3)),
treatment =c(rep("Control", 4), rep("Condicion1",4)))
head(df)
Es un dataframe con 3 variables(genes, expression y treatment) y cada uno tiene su propia clase.
str(df)
Dataframe con los datos de la clase#
Vamos a crear un nuevo vector con las edades, llamado edad, posteriormente, vamos a combinarlo con el vector que creamos previamente ClaseBioinfo
# declarar un vector e incorporarlo en el dataframe
edad <- c(31,32, 30, 30, 28, 32, 29)
# Almacenar el dataframe
ClaseBioinfo <- data.frame(ClaseBioinfo, edad)
ClaseBioinfo
Agregar una columna en el dataframe#
Tambien podemos agregar nuevas columnas usando el simbolo $ de la siguiente manera:
ClaseBioinfo$formacion <- c("medicina", "nutricion", "medicina",
"biologia", "medicina", "medicina",
"biologia")
head(ClaseBioinfo,3)
El dataframe tiene 7 filas y 3 columnas.
str(ClaseBioinfo)
Dataframe, completando espacios faltantes#
Cuando creamos la columna formacion en el dataframe al contener 9 elementos el dataframe, R repite 3 veces los valores contenidos en la columna formacion para llenar los 9 espacios.
ClaseBioinfo <- c("Jose Luis", "Diego", "Rosita", "Yuri",
"Ulises", "Tere", "Chucho", "Evelia", "Fulanito")
edad <-c(31,32, 30, 30, 28, 32, 29,29,30)
ClaseBioinfo <- data.frame(ClaseBioinfo, edad) # crear dataframe
ClaseBioinfo$formacion <- c("medicina", "nutricion", "medicina")
head(ClaseBioinfo, 3)
Ejercicios:#
A partir del siguiente ejemplo, completa los siguientes ejercicios:
df <- data.frame(genes = paste0("Gen", seq_len(8)),
expression = c(3.8, 5.5, 6.3, 1.8, 9, rep(3,3)),
treatment =c(rep("Control", 4), rep("Condicion1",4)))
- Convierte la columna 3 perteneciente a tratamiento (treatment) a factor.
NOTA: En este ejemplo, solo tenemos dos condiciones “Control” y “Condicion1”. Por lo que, solo deben hacer dos niveles (levels).
- Renombra las filas con los nombres de los genes y elimina la columna 1.
NOTA: Recuerda que para renombrar las filas es rownames() y las columnas se usa colnames().
Respuestas#
- Modifica la columna 3 perteneciente a tratamiento (treatment) a factor.
df$treatment <- as.factor(df$treatment) # conversion a factor
str(df)
NOTA: El signo de dinero $ nos permite seleccionar una columna (variable) de un dataframe.
- Renombra las filas con los nombres de los genes y elimina la columna 1.
rownames(df) <- df$genes # renombrar
df <- df[,-1] # eliminar columna 1
Index#
Por medio de un index podemos:
1) Obtener la información de un dato en específico.
2) Modificar un dato en específico.
3) Eliminarlo un dato en específico.
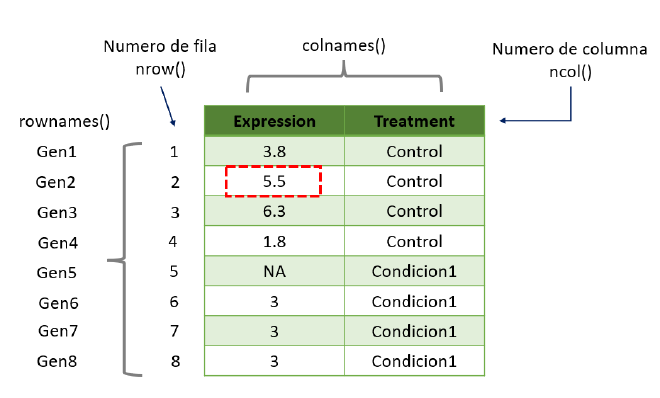
Podemos hacer la pregunta de dos maneras, 1) ¿Cuál es el nivel de expresión del Gen2? O 2) ¿Que gen contiene una expresión de 5.5?
Ejercicios#
- ¿Cuál es el nivel de expresión del Gen2?
NOTA: Recuerda que un dataframe tiene dos dimensiones 1) fila y 2) columna, dataframe[fila, columna]. Estos nos ayudaran como coordenadas en el dataframe.
# Opcion A
df[2,1]
# [1] 5.5
# Opcion B
df$expression[2]
# [1] 5.5
# Opcion C
df["Gen2",]
# expression treatment
# Gen2 5.5 Control
df["Gen2",1]
# [1] 5.5
# Opcion D
df["Gen2","expression"]
# [1] 5.5
- ¿Que gen contiene una expresión de 1.8?
# Opcion A
df[df[, "expression"] == 1.8,]
# expression treatment
# Gen4 1.8 Control
df[df[, 1] == 1.8,]
# expression treatment
# Gen4 1.8 Control
# Opcion B
df[df == 1.8,]
# expression treatment
# Gen4 1.8 Control
# Opcion C
df[df$expression == 1.8,]
# expression treatment
# Gen4 1.8 Control
# Opcion D
subset(df, expression == 1.8)
# expression treatment
# Gen4 1.8 Control
Index usando un vector#
Podemos crear una variable i que será nuestro index para extraer valores
x <- 1:5
i <- c(1, 3) # el index es numero y nos permite extraer la posicion 1 y 3 del vector
x[i]
Usar el vector i para obtener las filas 1 y 3
df[i,]
# expression treatment
# Gen1 3.8 Control
# Gen3 6.3 Control
NOTA: Recuerda que una matriz esta compuesta de filas y columnas.
Ejercicio 1 - Vector#
# Declarar el vector con 5 posiciones (del 1 al 5)
x <- 1:5
x
# Observar el componente / posicion 3
x[3]
# Observar multiles posiciones
x[c(1,3)]
x[c(1:3,5)] # OR x[-4]
# Sustituir el valor de la posicion 3
x[3] <- 20
x
# Eliminar la posicion 1
x[-1]
Ejercicio 2 - Matriz#
# Genera una matriz de tamano 2 x 3 (filas, columnas) (rows, columns)
x <- matrix(1:6, 2, 3)
x
# # Sustituir los valores presentes en la columna 3 de la matriz
x[, 3] <- 21:22
x
# Visualizacion en modo de matriz
x[, 3, drop = FALSE]
# Eliminar la columna 1
x[, -1]
# Eliminar multiples columnas y Visualizacion en modo de matriz
x[, -(1:2), drop = FALSE] # es lo mismo que x[, 3, drop = FALSE]
Ejercicio 3 - Lista#
# Opcion A
n=c(2, 3, 5)
s=c("aa", "bb", "cc", "dd", "ee")
x=list(n, s, 3)
x
# Opcion B
x <- list(c(2, 3, 5), c("aa", "bb", "cc", "dd", "ee"), 3)
x
Ejercicio 4 - Acceder a los elementos de la lista#
x[2] # 2do elemento, los datos con titulo
x[[2]] # 2do elemento, solo los datos
x[c(2, 3)] # 2ndo y 3er elemento
Acceder a un dato de un elemento
x[[c(2, 3)]] # dato 3 del elemento 2
Ejercicio 5 - Operaciones#
# Ejercicio 1
x <- 1:10 # Declarar un vector
x[x >= 5] <- 20 # todos los numeros que sean mayor o igual a 5 sustituir por 20
x
# A partir del resultado de x realiza lo siguiente
x[x == 1] <- 25 # todos los numeros que sean igual a 1 sustituir por 25
x
# Ejercicio 2
x <- matrix(1:20, 4, 5) #Genera una matriz de tamano 4 x 5 (filas, columnas) (rows, columns)
x # Visualizar la matriz
# A partir de la matriz generada realiza lo siguiente
x[, 5] <- 51:54 # 1. ¿Qué cambios existieron en la matriz?
x # Visualizar la matriz de salida
# 2. ¿Cuáles son los valores presentes en la columna 3 de la matriz?
x[, 5]
#3. ¿Como cambia el valor de salida cuando coloco drop= FALSE?
x[, 5, drop = FALSE]
Ejercicio 6 - Operaciones#
# Ejercicio 3.
# 1. ¿Como cambia el valor de salida al eliminar la columna 3?
x <- matrix(1:16, 4, 4) #Genera una matriz de tamano 4 x 5 (filas, columnas) (rows, columns)
x[, -1]
# 2. ¿Como cambia el valor de salida cuando coloco drop= FALSE?
x[, -(1:2), drop = FALSE]
# 3. Renombra las filas y columnas empleando rownames() y colnames(). En fila coloca los nombres: Gen 1, Gen 2, Gen 3 y Gen 4. Y en columnas coloca los nombres: SRA_1,SRA_2 Y SRA_3
# Ejercicio 4. Realiza las siguientes operaciones
# 1. Sumatoria
x <- 1:4
y <- 1:2
z <- x + y
z
# 2. Multiplicacion
x <- 1:4
a <- 10
z <- a * x
z
Fuentes (Viernes 2):
Viernes de Bioinformatica en el LIIGH, 2 febrero - 2 de agosto 2024


