La manipulación de datos con funciones de R base es un tema esencial en análisis de datos. En este contexto, R ofrece una variedad de herramientas para transformar, filtrar y resumir datos. Desde la importación hasta la limpieza, estas funciones te permiten trabajar con data frames y vectores de manera eficiente.
La manipulación de datos implica tareas como filtrar filas, seleccionar columnas, crear nuevas variables y calcular estadísticas descriptivas. Además, puedes combinar múltiples data frames y aplicar operaciones a nivel de fila o columna.
Paquetes necesarios#
install.packages("dplyr") # Manipulación de datos
install.packages("tidyr") # Manipulación de datos
install.packages("tidyverse")# Manipulación de datos
install.packages("reshape2") # Transformación de datos
install.packages("ggplot2") # Visualización grafica
install.packages("cowplot") # Generar varios graficos en una misma figura
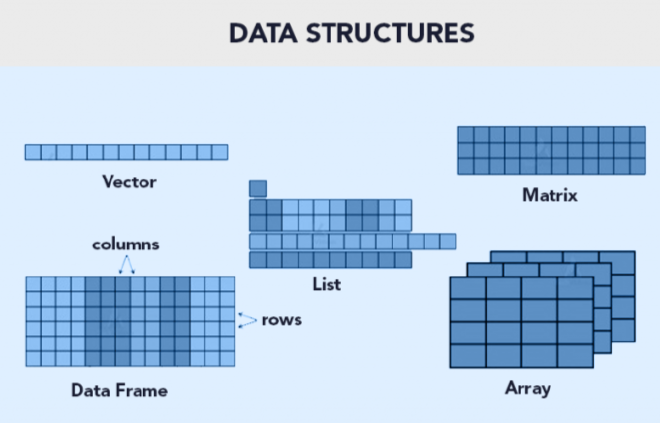
Funciones básicas en R#
| Función | Empleo | Documentación | Empleos |
|---|---|---|---|
| sum() | Adición / suma | ?sum | sum(2+7) |
| prod() | Sustracción/resta | ?prod | prod(9-2) |
| max() | Valor máximo | ?max | max(c(1,2,3,4,5)) |
| min() | Valor mínimo | ?min | min(1:5) |
| range() | Rango | ?range | range(c(2,8,7,6,1)) |
| mean() | Promedio | ?mean | mean(c(2,8,7,6,1)) |
| median() | Mediana | ?median | median(c(2,8,7,6,1)) |
| var() | Varianza | ?var | var(1:10); var(1:5, 1:5) |
| con() | Covarianza | ?cov | cov(1:10, 2:11) |
| cor() | Matriz de correlación | ?cor | cor(1:10,2:11) |
| unique() | Valor unico | ?unique | unique(df$expression) |
| sort() | Ordenar | ?sort | sort(df$expression) |
| order() | Ordenar | ?order | df[order(df$expression),] |
| table() | cuentas de cada valor en tablas | ?table | table(df) |
| subset() | Extraer información de una MATRIZ o DATAFRAME | ?subset | subset(df, expression == 1.8) |
| sample() | Extraer información de manera aleatoria | ?sample | sample(df$expression, size = 3) |
| length() | Número de elementos de un VECTOR | ?length | length(df$expression) |
| dim() | Dimensiones de una MATRIZ o DATAFRAME | ?dim | dim(df) |
| is.na() | Es un valor NA? | ?is.na | is.na(df) |
| t() | Cambiar filas por columnas (transposición) | ?t | t(df) |
Existen otras funciones matematicas como log, exp,log10, log2, sin, cos, tan, asin, acos, atan, abs, sqrt, etc.
Otras funciones importantes son rbind() y cbind()#
Puedes encontrar su documentación empleando ?rbind y ?cbind
cbind()#
Podemos unir las columnas dos vectores.
a <- c(1, 3, 3, 4, 5)
b <- c(7, 7, 8, 3, 2)
new_matrix <- cbind(a, b)
new_matrix
# a b
# [1,] 1 7
# [2,] 3 7
# [3,] 3 8
# [4,] 4 3
# [5,] 5 2
Crear un dataframe
df <- data.frame(genes = paste0("Gen", seq_len(8)),
expression = c(3.8, 5.5, 6.3, 1.8, 9, rep(3,3)),
treatment =c(rep("Control", 4), rep("Condicion1",4)))
Tambiend podemos incorporarle una nueva columna a un dataframe
date_sample <- rep(c(11, 12, 13, 14),2)
df <- cbind(df, date_sample)
# Renombrar columna 4
colnames(df)[4] <- "Date"
head(df)
# genes expression treatment Date
# 1 Gen1 3.8 Control 11
# 2 Gen2 5.5 Control 12
# 3 Gen3 6.3 Control 13
# 4 Gen4 1.8 Control 14
# 5 Gen5 9.0 Condicion1 11
# 6 Gen6 3.0 Condicion1 12
rbinf()#
Vamos a unir dos dataframe pero ambos deben tener el mismo numero y nombre en su columnas.
NOTA: Los rownames deben ser Únicos, por lo que no debemos comenzar desde el Gen1.
df_v2 <- data.frame(genes = paste0("Gen", 9:16),
expression = c(6.1, 6.2, 5, 2, 7, rep(8,3)),
treatment = c(rep("Control", 4), rep("Condicion1",4)),
Date = rep(c(11, 12, 13, 14),2) )
df_v2$treatment <- as.factor(df_v2$treatment) # conversión a factor
#unir
df_new <- rbind(df, df_v2)
head(df_new)
# genes expression treatment Date
# 1 Gen1 3.8 Control 11
# 2 Gen2 5.5 Control 12
# 3 Gen3 6.3 Control 13
# 4 Gen4 1.8 Control 14
# 5 Gen5 9.0 Condicion1 11
# 6 Gen6 3.0 Condicion1 12
Información de la estructura de un dataframe#
Para ver su estructura e información:
dim(df) # dimensiones [fila, columna]
length(df) # largo, número de columnas
ncol(df) # número de columnas
nrow(df) # número de filas
names(df) # nombre de las columnas
str(df) # Estructura
Podemos adornar la salida colocando estas notas.
cat("Dimensiones:", dim(df), "\n")
# Dimensiones: 8 4
cat("Numero de columnas:", ncol(df), "\n")
# Numero de columnas: 4
cat("Numero de filas:", nrow(df), "\n")
# Numero de filas: 8
cat("Nombre de las columnas:", names(df), "\n")
# Nombre de las columnas: genes expression treatment Date
NOTA: El “\n” indica saltos de linea en el texto.
Función which()#
Podemos usar esta función para extraer la informacion y/o las posiciones que cumpla con un argumento logico.
Documentación ?which
x <- c(1, 5, 4, 8, 4)
which(x == 4 | x == 1) # condicional que sea igual a 4 o a 1
# [1] 1 3 5
¿Cuántos números son iguales a 4 y a 1?
length(which(x == 4 | x == 1))
# [1] 3
En una matriz#
Ejemplo 1: Obtener las valores que al divirse entre 3 sean iguales a cero#
m <- matrix(1:12, 3, 4) # Generar una matriz de 3 x 4
# Opción A - con R base
m[m %% 3 == 0]
# [1] 3 6 9 12
# Opción B - con la función which
div.3 <- m %% 3 == 0
which(div.3) # OR which(m %% 3 == 0)
# [1] 3 6 9 12
Ejemplo 2: Obtener las posiciones de los numeros que al divirse entre 3 sean iguales a cero#
# Para obtener la información de las posiciones podemos usar:
which(div.3, arr.ind = TRUE)
# row col
# [1,] 3 1
# [2,] 3 2
# [3,] 3 3
# [4,] 3 4
# Ejemplo 2
rownames(m) <- paste("Case", 1:3, sep = "_") # renombrar las filas
which(m %% 5 == 0, arr.ind = TRUE) # Extraer las posiciones que cumplan con la condición logica
# row col
# Case_2 2 2
# Case_1 1 4
Index#
Por medio de un index podemos:
1) Obtener la información de un dato en específico.
2) Modificar un dato en específico.
3) Eliminarlo un dato en específico.
Podemos hacer la pregunta de dos maneras, 1) ¿Cuál es el nivel de expresión del Gen2? O 2) ¿Que gen contiene una expresión de 5.5?
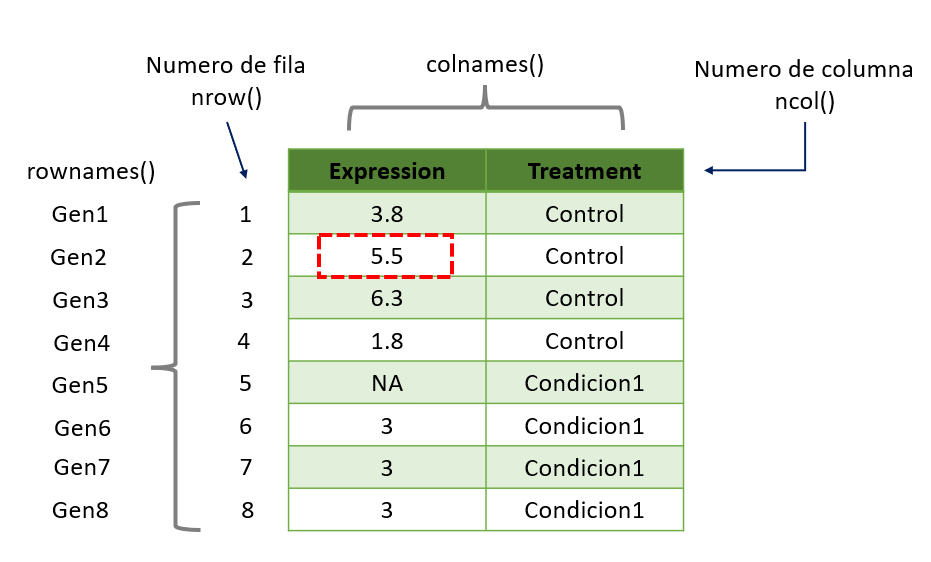
Función which() en un dataframe#
¿Que gen contiene una expresión de 1.8?
# Opción A
which(df == 1.8, arr.ind = TRUE)
# row col
# [1,] 4 2
# Opción B
df[which(df$expression == 1.8), ]
# genes expression treatment Date
# 4 Gen4 1.8 Control 14
Repaso#
Paso 1. Crear un RBioProject#

Paso 2. Descargar los datos y verificar su ubicaciónen la carpeta data/#
Descarga los tres datasets de Marvel dando click en este link, debes tener tres archivos en la misma carpeta
charactersStats.csv,heroesInformation.csvysuperHeroPowers.csv. Asegúrate de guardarlo en el directorio donde hiciste tu Rproject. En mi caso, guarde los datos en la carpetadata/.Verifica que se encuentren los tres archivos en la carpeta
data/.
dir("data")
# [1] "charactersStats.csv" "dirty_iris.csv" "fullMarvelDc.RData"
# [4] "heroesInformation.csv" "Info_data.RData" "marvelDc.RData"
# [7] "marvelDcInfo.RData" "MMHOME.csv" "planets.csv"
# [10] "superheroes.RData" "superHeroPowers.csv"
Paso 3. Importar los datos en R#
- Importa los archivos en R de la siguiente manera:
infoCharacters <- read.csv("data/heroesInformation.csv", na.strings = c("-", "-99"))
# La opción na.string nos permite sustituir valores - y -99 por NA
infoPowers <- read.csv("data/superHeroPowers.csv")
infoStats <- read.csv("data/charactersStats.csv", na.strings = "")
Visualiza su información con head() o View().
NOTA: Primero debes crear la carpeta data/ antes de descargar los archivos.
Buenas prácticas#
Nuestro script debe verse asi:
######
# Script de clase "Intro a R"
# Evelia Coss
# 8 de marzo 2024
#######
indir = "C:/Users/ecoss/OneDrive - CINVESTAV/Documentos/Posdoc_LIIGH/VieRnesBioinfo/ViernesBioinfo_2024/Presentaciones/data/"
outdir = "C:/Users/ecoss/OneDrive - CINVESTAV/Documentos/Posdoc_LIIGH/VieRnesBioinfo/ViernesBioinfo_2024/Presentaciones/"
# Importar datos
infoCharacters <- read.csv(paste0(indir,"heroesInformation.csv"), na.strings = c("-", "-99"))
# La opción na.string nos permite sustituir valores - y -99 por NA
infoPowers <- read.csv(paste0(indir,"superHeroPowers.csv"))
infoStats <- read.csv(paste0(indir,"charactersStats.csv"), na.strings = "")
Paso 4. Renombrar la columna Name en todos los dataframe#
Vamos a unificar el nombre las columnas que tienen los nombre en todas las columnas colocando el nombre Name. El dataframe infoStats ya contiene ese nombre en la columna 1.
colnames(infoCharacters)[colnames(infoCharacters) == "name"] <- "Name"
colnames(infoPowers)[colnames(infoPowers) == "hero_names"] <- "Name"
Paso 5. Seleccionar SOLO los datos de Marvel Comics y DC Comics#
Podemos usar la función unique() para obtener los valores únicos en un vector o de una columna en un dataframe.
# Empresas comprendidas en esta base de datos
unique(infoCharacters$Publisher)
# [1] "Marvel Comics" "Dark Horse Comics" "DC Comics"
# [4] "NBC - Heroes" "Wildstorm" "Image Comics"
# [7] "" "Icon Comics" "SyFy"
# [10] "Hanna-Barbera" "George Lucas" "Team Epic TV"
# [13] "South Park" "HarperCollins" "ABC Studios"
# [16] "Universal Studios" "Star Trek" "IDW Publishing"
# [19] "Shueisha" "Sony Pictures" "J. K. Rowling"
# [22] "Titan Books" "Rebellion" "Microsoft"
# [25] "J. R. R. Tolkien"
Verificamos las dimensiones#
Esperariamos que cada fila sea un solo personaje, pero este dataframe contiene nombres repetidos.
dim(marvelDcInfo)
# [1] 603 11
Esperariamos que las filas representaran el numero de personales, teniendo un total de 603 personajes y las columnas representaran las variables, teniendo un total de 11 variables.
Sin embargo, podemos extraer los nombres de los personajes que solo se repitan una vez y veremos que nuestro dataset no esta limpio. Ya que solo tiene un total de 585 personajes.
length(unique(marvelDcInfo$Name))
# [1] 585
Observar valores duplicados#
Observar cuales se duplican mediante la función duplicated() de R base.
head(marvelDcInfo[duplicated(marvelDcInfo$Name), ], 3)
# X Name Gender Eye.color Race Hair.color Height Publisher
# 49 48 Atlas Male blue God / Eternal Brown 198 DC Comics
# 51 50 Atom Male <NA> <NA> <NA> -99 DC Comics
# 64 63 Batgirl Female green Human Red 170 DC Comics
# Skin.color Alignment Weight
# 49 <NA> bad 126
# 51 <NA> good -99
# 64 <NA> good 57
Para saber cuantos personajes duplicados podemos usar length()
length(marvelDcInfo[duplicated(marvelDcInfo$Name), ])
# [1] 11
Existen 11 personajes que se encuentran duplicados en esta base de datos.
Revisemos un ejemplo de datos duplicados#
Vamos a obtener la información de Batman y vemos que tiene dos filas pertenecientes al mismo personaje.
# Opción A
marvelDcInfo[marvelDcInfo$Name == "Batman", ]
# X Name Gender Eye.color Race Hair.color Height Publisher Skin.color
# 69 68 Batman Male blue Human black 188 DC Comics <NA>
# 70 69 Batman Male blue Human Black 178 DC Comics <NA>
# Alignment Weight
# 69 good 95
# 70 good 77
Tambien podemos usar la función subset() para extraer información de algun personaje.
# Opción B
subset(marvelDcInfo, Name == "Batman")
# X Name Gender Eye.color Race Hair.color Height Publisher Skin.color
# 69 68 Batman Male blue Human black 188 DC Comics <NA>
# 70 69 Batman Male blue Human Black 178 DC Comics <NA>
# Alignment Weight
# 69 good 95
# 70 good 77
Empecemos con la limpieza de los datos#
Paso 6. Eliminar duplicados#
El signo de exclamación ! nos permite indicar lo contrario, es decir, devolverme todos los que no esten duplicados seria asi !duplicated().
marvelDcInfo <- marvelDcInfo[!duplicated(marvelDcInfo$Name), ]
Paso 7. Seleccionar columnas#
marvelDcInfo <- marvelDcInfo[, c("Name", "Gender", "Race", "Publisher")]
head(marvelDcInfo, 3)
# Name Gender Race Publisher
# 1 A-Bomb Male Human Marvel Comics
# 3 Abin Sur Male Ungaran DC Comics
# 4 Abomination Male Human / Radiation Marvel Comics
Paso 8. Cambiar formatos en algunas columnas#
Debemos convertir las columnas Name, Gender, Race y Publisher de character a factor.
str(marvelDcInfo)
# 'data.frame': 585 obs. of 4 variables:
# $ Name : chr "A-Bomb" "Abin Sur" "Abomination" "Abraxas" ...
# $ Gender : chr "Male" "Male" "Male" "Male" ...
# $ Race : chr "Human" "Ungaran" "Human / Radiation" "Cosmic Entity" ...
# $ Publisher: chr "Marvel Comics" "DC Comics" "Marvel Comics" "Marvel Comics" ...
Para cambiar a factor usamos la función as.factor().
marvelDcInfo$Name <- as.factor(marvelDcInfo$Name)
marvelDcInfo$Gender <- as.factor(marvelDcInfo$Gender)
marvelDcInfo$Race <- as.factor(marvelDcInfo$Race)
marvelDcInfo$Publisher <- as.factor(marvelDcInfo$Publisher)
Ejercicios#
¿Cuántos personajes hay por cada empresa?
¿Cuántos personajes son mujeres y hombres hay por cada empresa?
¿Cuántas razas hay en el dataframe?
¿Cuáles son las razas predominantes de cada empresa?
1) ¿Cuántos personajes hay por cada empresa?#
Opcion A#
summary(marvelDcInfo$Publisher)
# DC Comics Marvel Comics
# 206 379
Opcion B#
table(marvelDcInfo$Publisher)
#
# DC Comics Marvel Comics
# 206 379
2) ¿Cuántos personajes son mujeres y hombres hay por cada empresa?#
DC Comics#
cat("DC Comics, hombres:", nrow(subset(marvelDcInfo, Publisher == "DC Comics" & Gender == "Male")), "\n")
# DC Comics, hombres: 147
cat("DC Comics, mujeres:", nrow(subset(marvelDcInfo, Publisher == "DC Comics" & Gender == "Female")), "\n")
# DC Comics, mujeres: 58
Marvel#
cat("Marvel, hombres:", nrow(subset(marvelDcInfo, Publisher == "Marvel Comics" & Gender == "Male")), "\n")
# Marvel, hombres: 245
cat("Marvel, mujeres:", nrow(subset(marvelDcInfo, Publisher == "Marvel Comics" & Gender == "Female")), "\n")
# Marvel, mujeres: 110
3) ¿Cuántas razas hay en el dataframe?#
Opción A#
Eliminamos los NA que tengamos con !is.na(), posteriormente, obtenemos los nombres unicos y medimos.
length(unique(marvelDcInfo$Race[!is.na(marvelDcInfo$Race)]))
# [1] 48
Opción B#
Como esta columna se encuentra en factores, podemos ver el numero de niveles con a funcion nlevels().
nlevels(marvelDcInfo$Race)
# [1] 48
4) ¿Cuáles son las razas predominantes de cada empresa?#
Podemos usar la función sort() para ordenar los datos de mayor a menor, usando el argumento decreasing = TRUE.
La raza Human o humana es la mas predominante en ambas empresas, seguida de la mutante en el caso de Marvel Comics.
head(sort(table(marvelDcInfo$Race), decreasing = TRUE), 10)
#
# Human Mutant Human / Radiation God / Eternal
# 167 58 11 10
# Symbiote Alien Kryptonian Android
# 8 7 7 6
# Cyborg Asgardian
# 6 5
Guardar variables para la siguiente clase#
Guardar una sola variable
save(marvelDcInfo, file = "Presentaciones/data/marvelDcInfo.RData")
Guardar varias variables
save(infoStats, infoPowers, marvelDcInfo, file = "Presentaciones/data/Info_data.RData")

